LDA
Nov 27, 2020 21:13 · 590 words · 3 minute read
library(MASS)
wais <- read.csv('WAIS-data-Table22-12.csv')[,-2]
head(wais)
## IGROUP INFORMATION SIMILARITIES ARITHMETIC PICTURE
## 1 2 9 5 10 8
## 2 2 10 0 6 2
## 3 2 8 9 11 1
## 4 2 13 7 14 9
## 5 2 4 0 4 0
## 6 2 4 0 6 0Data description
WAIS Data: Wechsler Adult Intelligence Scale for two categories of people
group1: people who do not present a senile factor(노인성 인자)
group2: people who do present a senile factor
| 변수명 | 데이터 타입 | 변수 설명 |
|---|---|---|
| X1 | NUM | Information |
| X2 | NUM | Similarities |
| X3 | NUM | Arithmetic |
| X4 | NUM | Picture completion |
group1은 노인성 인자를 보이지 않는 그룹이고 group2는 노인성 인자를 보인은 그룹이다. 현재 WAIS에 따라 각 피험자에 대한 4가지 지표로 표현된 데이터를 구하였다. 이 때, 이 4가지 지표(X1,⋯,X4)를 기준으로 IGROUP 1,2가 나뉘었을 때 이 분류를 가장 잘 나타내는 선은 무엇일까? 현재 독립변수를 INFORMATION, SIMILIARITIES, ARITHMETIC, PICTURE로 잡고 종속변수를 IGROUP으로 판단할 수 있다. 그리고 이 상황에서 LDA(Linear Discriminant Analysis)를 통하여 regrouping 후 실제 분류값과 비교할 것이다. LDA는 그룹내분산의 최소화, 그룹간거리의 최대화를 기준으로 선을 긋는다. 그룹이 2개로 나누어져있으므로 판별기준은 선으로 나타난다.
Analysis
현재 데이터를 이용해 알 수 있는 sample prior probability는 3749(group1), 1249(group2)이다. 그리고 실제 LDA를 한 결과는 다음과 같다.
(ld.wais <- lda(formula = IGROUP~. , data=wais))
## Call:
## lda(IGROUP ~ ., data = wais)
##
## Prior probabilities of groups:
## 1 2
## 0.755102 0.244898
##
## Group means:
## INFORMATION SIMILARITIES ARITHMETIC PICTURE
## 1 12.56757 9.567568 11.48649 7.972973
## 2 8.75000 5.333333 8.50000 4.750000
##
## Coefficients of linear discriminants:
## LD1
## INFORMATION -0.016892707
## SIMILARITIES -0.132789096
## ARITHMETIC -0.005504696
## PICTURE -0.285326128
apply(ld.wais$means %*% ld.wais$scaling, 2, mean)
## LD1
## -3.039502계수는 다음과 같이 나타난다.
-0.017(INFO), -0.133(SIM), -0.006(ARTH), -0.285(PICT)
계수는 lda로 계산된 식의 계수를 의미하며. 여기서는 1,2로 두개의 범주가 나눠져있으므로 ld1만 나타난다. apply를 통해 계산한 값은 판별함수의 상수항이며 계수에 평균값을 실제 대입하면 얻을 수 있는 값이다.
library(tibble)
REGROUP = predict(ld.wais, wais)$class
wais <- add_column(wais, REGROUP, .after='IGROUP')
wais$IGROUP <- as.factor(wais$IGROUP)
head(wais)
## IGROUP REGROUP INFORMATION SIMILARITIES ARITHMETIC PICTURE
## 1 2 1 9 5 10 8
## 2 2 2 10 0 6 2
## 3 2 2 8 9 11 1
## 4 2 1 13 7 14 9
## 5 2 2 4 0 4 0
## 6 2 2 4 0 6 0wais의 데이터를 실제 LDA를 통해 regrouping한 결과를 열에 추가하였다. 그렇다면 실제 IGROUP과 REGROUP은 얼마만큼의 차이가 있을까?
library(GGally)
## 필요한 패키지를 로딩중입니다: ggplot2
## Registered S3 method overwritten by 'GGally':
## method from
## +.gg ggplot2
ggpairs(wais[,-2], aes(colour=IGROUP))
## `stat_bin()` using `bins = 30`. Pick better value with `binwidth`.
## `stat_bin()` using `bins = 30`. Pick better value with `binwidth`.
## `stat_bin()` using `bins = 30`. Pick better value with `binwidth`.
## `stat_bin()` using `bins = 30`. Pick better value with `binwidth`.
ggpairs(wais[,-1], aes(colour=REGROUP))
## `stat_bin()` using `bins = 30`. Pick better value with `binwidth`.
## `stat_bin()` using `bins = 30`. Pick better value with `binwidth`.
## `stat_bin()` using `bins = 30`. Pick better value with `binwidth`.
## `stat_bin()` using `bins = 30`. Pick better value with `binwidth`.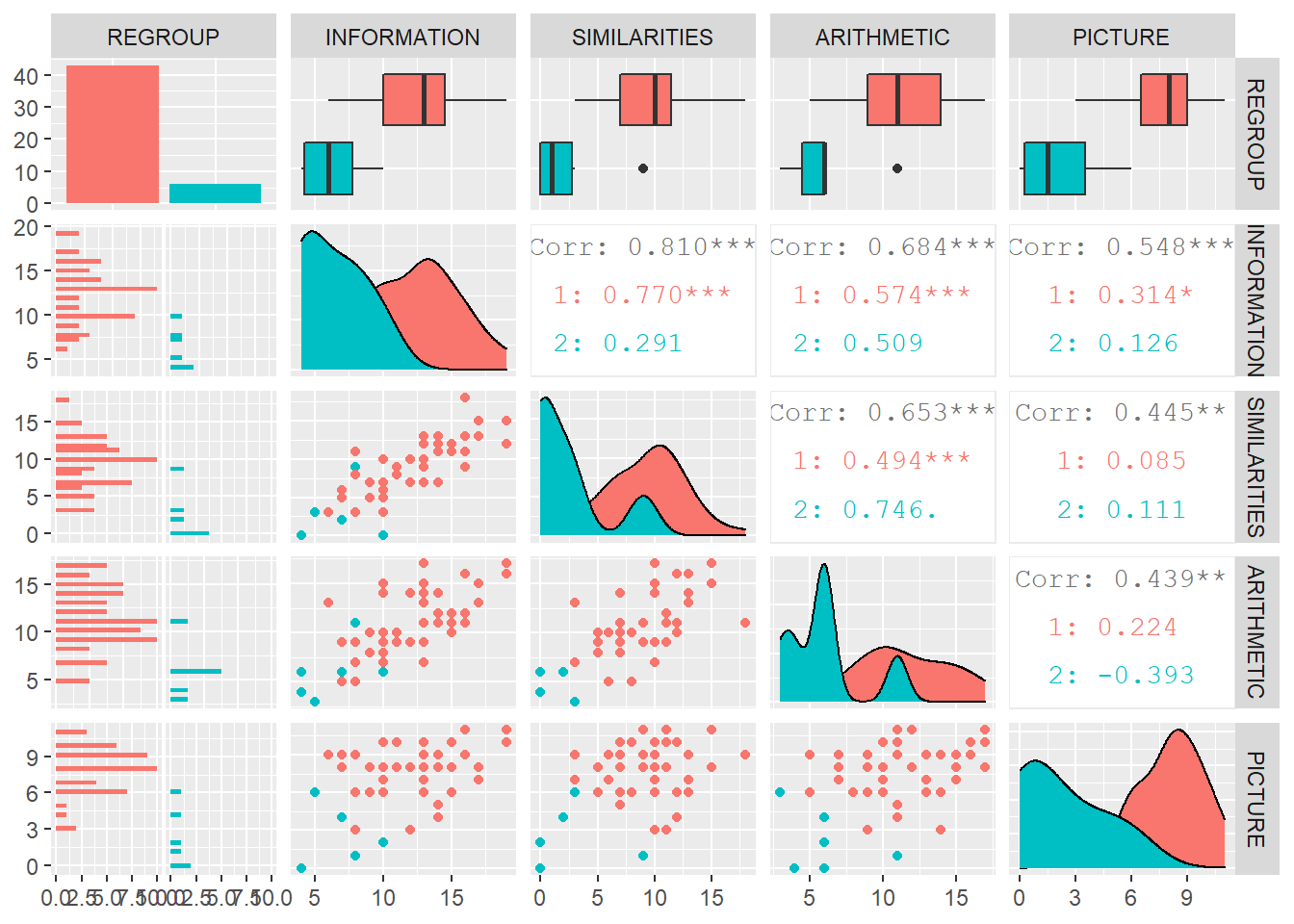
table(wais[,1], wais[,2])
##
## 1 2
## 1 37 0
## 2 6 6위의 플랏은 IGROUP을 기준으로 색이 나뉜 것이고, 밑의 플랏은 REGROUP을 기준으로 색을 나눈 것이다.
테이블에서 보이듯이 정분류는 43개이며, 오분류는 6개이다.
정분류는 1을 1로 분류한 것이 36개, 2를 2로 분류한 게 6개이다.
그리고 igroup의 2를 regroup에서 1로 오분류 한것이 6개로 나타난다. 오분류의 확률은 649로 나타난다.
plot(ld.wais)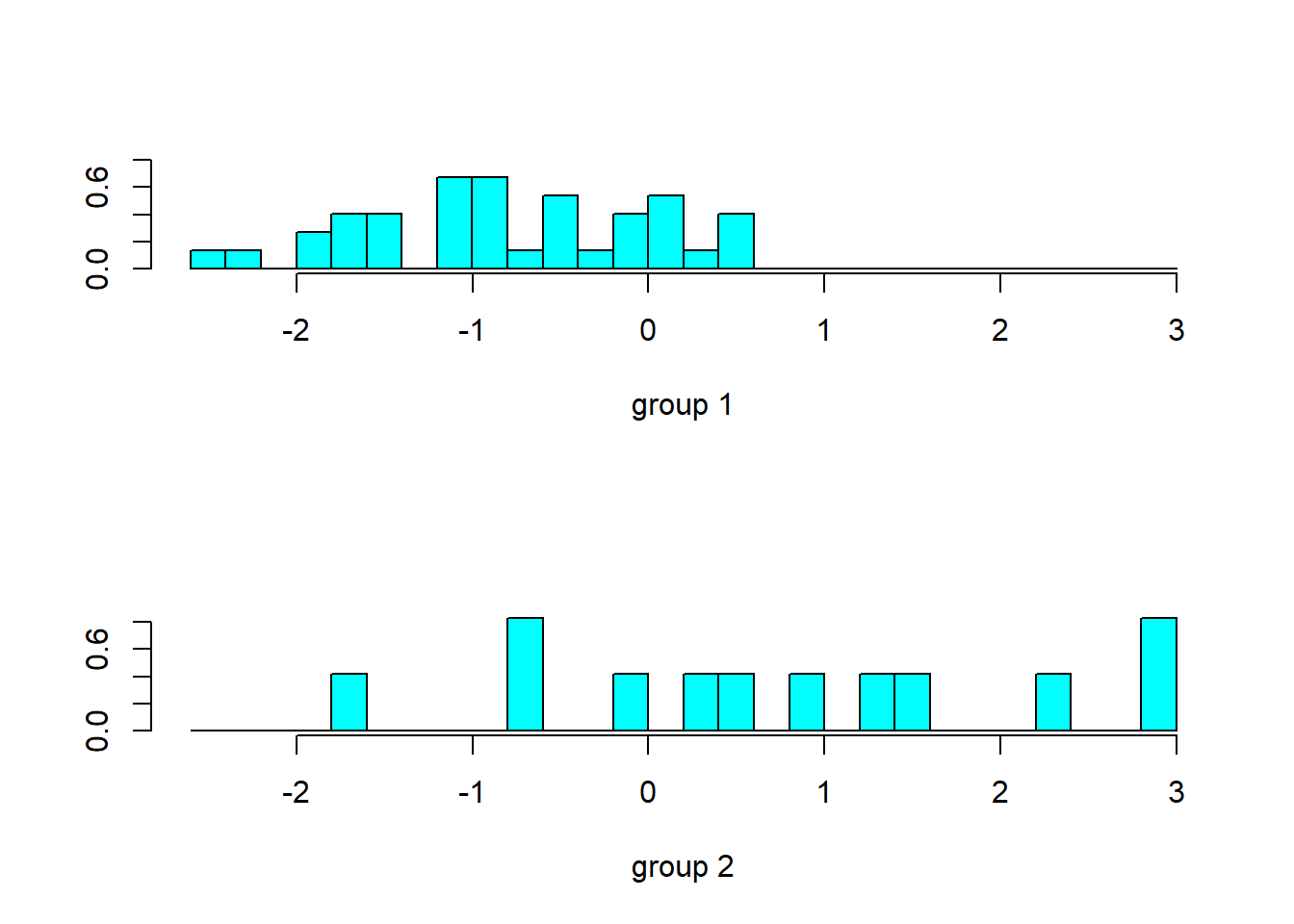
위의 그래프는 각 그룹의 데이터가 new axis에 projection된 후의 분포를 나타낸다. 실제 group1과 group2는 new axis의 0을 기준으로 어느정도 판별이 되고 있음을 보인다.